Das Projekt:
Chlorophyll-a-Satellitendaten (http://oceancolor.gsfc.nasa.gov/) sollen so aufbereitet werden, dass sie dieselbe räumliche Struktur aufweisen wie die aus einem Ökosystemmodell (ECOHAM) hervorgehenden simulierten Chlorophyll-a-Daten (http://www.ifm.uni-hamburg.de/~wwwem/em.html). Die Anpassung der räumlichen Abbildungsstruktur und die Aufbereitung der vorliegenen Daten sollen für einen späteren Vergleich zwischen Satelliten- und Modelldaten genutzt werden.
Die Teilziele des Projektes:
- Herunterladen und Speichern der Satellitendaten
- Anpassen der Satellitendaten an die Modellgitterstruktur und an das Modellgebiet (Nordseeregion)
- graphische Darstellung mit GMT
- Schließung der Lücken in den Satellitendaten durch räumliche und zeitliche Interpolation
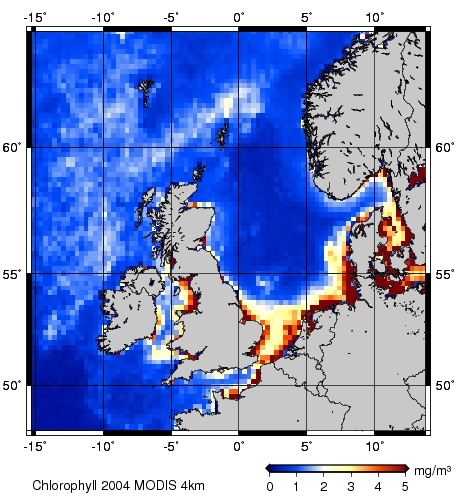
Ergebnisbeispiel: Die nachfolgende Abbildung zeigt die vom Satelliten gemessenen Chlorophyll-a-Werte gemittelt über das Jahr 2004. Die Satellitendaten sind auf das Modellgebiet (North European Continental Shelf) zu geschnitten und so interpoliert, dass, wie im Modell, 88 Werte in der x-Richtung und 82 Werte in der y-Richtung vorhanden sind.
Beteiligte:
Dennis Brüning, Lars Kaleschke, Nina Maaß, Markéta Pokorná, Bente Tiedje
Projektleiterin: Bente Tiedje
Projektplanung:
Für das Projekt wurden 2 1/2 Tage geplant und im Blockseminar reserviert.
- Montag:
- Einführung in das Thema
- Besprechung und Festlegung der Teilziele
- Einteilung in Arbeitsgruppen
- Beginn der Gruppenarbeit
- Vorstellung der Gruppenarbeit: Jede Gruppe erklärt am Ende des Tages ihren Programmcode (auch wenn er noch unvollständig ist), Problembesprechung
- Dienstag:
- Fortführung der Gruppenarbeit
- Am Ende des Tages : Vorstellung der Gruppenarbeit und Besprechung
- Abschluss der Gruppenarbeit (sofern die Teilziele in den Gruppen erreicht worden sind)
- 1/2 Mittwoch:
- Zusammenfügen der Teilergebnisse aus den Arbeitsgruppen und Testen des Endergebnisses
- Abschlussbesprechung
Python-Programmcodes:
Ergebnisse der Gruppenarbeit:
Für die Zukunft: ChlorophyllProgrammcodes
