|
Size: 971
Comment:
|
Size: 3594
Comment:
|
| Deletions are marked like this. | Additions are marked like this. |
| Line 11: | Line 11: |
| Wir haben ein Programm geschrieben um über ein großes Bild einen Box-Filter laufen zu lassen, der Mittelwert und Standardabweichungen in einem 2-dimensionalen Array zurückgibt. | Wir haben ein Programm geschrieben, um über ein großes Bild einen Box-Filter laufen zu lassen, der Mittelwert und Standardabweichungen in einem 2-dimensionalen Array zurückgibt. |
| Line 27: | Line 27: |
Weiter haben wir uns damit beschäftigt, das Originalbild mit dem "read_asar_imp" einzulesen und in db-Darstellung auszugeben. {{{#!python # Einlesen des Originalbildes file='/pf/u/u242027/SAR_raw/ASA_IMP_1PNDPA20060617_043346_000000162048_00362_22460_2136.N1' I=read_asar_imp(file) # Darstellung des ASAR-Bildes in db (image1.png) w=8 # Größe der "Unterboxen" A=mean_std_box(I,w) figure(2) imshow(10*log10(A[:,:,0]),vmin=-20,vmax=0,interpolation='nearest',origin='lower') }}} {{attachment:image1.png}} Probleme hatten wir bei der Reduzierung des Speckles. Es wurde daher beschlossen diesen Schritt am Mittwoch zunächst in der ganzen Gruppe zu besprechen und dann durchzuführen. === Mittwoch === Wir haben eine Klassifizierung nur über den Mittelwert durchgeführt. Dazu haben wir das Bild in db umgerechnet und uns ein Histogramm erzeugt, bei dem jedoch keine Klassen zu unterscheiden waren. Auch eine Filterung mit dem Lee-Filter und dem Programm smooth hat am Histogramm nicht so viel verändert. Daher haben wir aus dem gemittelten Bild homogene Bereiche ausgewählt und diese in einem Histogramm geplottet. Insgesamt haben wir fünf Klassen unterschieden, wobei die fünfte Klasse (magenta) äußerst selten vorkam, aber einen sehr hohen Rückstreukoeffizienten hatte. {{{#!python def db_hist(I,b,r): """Input: I ist das Intensitätsbild, b ist die Anzahl der bins, r ist die Limitierung der x-Achse""" A_db=nan_to_num(10*log10(I)) h=histogram(A_db,bins=b,range=r,normed=True) return h[0],h[1] # h[0]: y-Achse des Histogramms, h[1]: x-Achse des Histogramms # Definition der homogenen Bereiche A_db=10*log10(A[:,:,0]) A1=A[295:394,929:997,0] A2=A[198:225,168:197,0] A3=A[863:949,382:464,0] A4=A[846:851,623:629,0] Aice=A[763:771,326:329,0] # vermutlich Eisberge b=50 r=[-20,5] y1,x1=db_hist(A1,b,r) y2,x2=db_hist(A2,b,r) y3,x3=db_hist(A3,b,r) y4,x4=db_hist(A4,b,r) yice,xice=db_hist(Aice,b,r) # Histogramm figure(7) plot(x1,y1,'g',x2,y2,'b',x3,y3,'y',x4,y4,'r',xice,yice,'m') }}} {{attachment:image2.png}} Die Schwellwerte haben wir aus dem Histogramm abgelesen. {{{#!python # Klassifikation # Schwellwerte s0=-17.3 s1=-12.4 s2=-7.88 s3=-1.73 A_class=A_db.copy() A_class[A_db<s0]=0 A_class[A_db>s0]=1 A_class[A_db>s1]=2 A_class[A_db>s2]=3 A_class[A_db>s3]=4 figure(11) imshow(A_class,cm.gist_rainbow,interpolation='nearest',origin='lower') colorbar() show() }}} {{attachment:image3.png}} Diese Bilder stimmen noch nicht ganz mit dem überein, was im Programmtext steht. Morgen geht es weiter. |
Besprechung ihrer Aufgaben ergab:
- Einlesen als Teilaufgabe wurde von Lars geloest Gruppe arbeitet direkt mit Bildkoordinaten, welche als Arrayindex dienen
Festlegung der Klasseneinteilung erst bei Bearbeitung der Aufgabe -> Sinnigkeitsentscheid Verwendung von Filtern und Clusterbasierte Merkmalsanalyse Ergebnis: die Zuordnung von Klassen zu Bildkoordinaten (x,y)
Dienstag
Wir haben ein Programm geschrieben, um über ein großes Bild einen Box-Filter laufen zu lassen, der Mittelwert und Standardabweichungen in einem 2-dimensionalen Array zurückgibt.
1 def mean_std_box(I,w):
2 """ Mittelwert über wxw Pixel"""
3 Y,X=I.shape # Einlesen der Bilddimension
4 M=zeros((Y/w,X/w,2),float)
5 a=range(0,Y/w,1)
6 b=range(0,X/w,1)
7 for y in a:
8 for x in b:
9 box=I[y*w:y*w+w-1,x*w:x*w+w-1]
10 M[y,x,0]=mean(box.flatten())
11 M[y,x,1]=std(box.flatten())
12 return M
Weiter haben wir uns damit beschäftigt, das Originalbild mit dem "read_asar_imp" einzulesen und in db-Darstellung auszugeben.
1 # Einlesen des Originalbildes
2 file='/pf/u/u242027/SAR_raw/ASA_IMP_1PNDPA20060617_043346_000000162048_00362_22460_2136.N1'
3 I=read_asar_imp(file)
4
5 # Darstellung des ASAR-Bildes in db (image1.png)
6 w=8 # Größe der "Unterboxen"
7 A=mean_std_box(I,w)
8
9 figure(2)
10 imshow(10*log10(A[:,:,0]),vmin=-20,vmax=0,interpolation='nearest',origin='lower')

Probleme hatten wir bei der Reduzierung des Speckles. Es wurde daher beschlossen diesen Schritt am Mittwoch zunächst in der ganzen Gruppe zu besprechen und dann durchzuführen.
Mittwoch
Wir haben eine Klassifizierung nur über den Mittelwert durchgeführt. Dazu haben wir das Bild in db umgerechnet und uns ein Histogramm erzeugt, bei dem jedoch keine Klassen zu unterscheiden waren. Auch eine Filterung mit dem Lee-Filter und dem Programm smooth hat am Histogramm nicht so viel verändert.
Daher haben wir aus dem gemittelten Bild homogene Bereiche ausgewählt und diese in einem Histogramm geplottet. Insgesamt haben wir fünf Klassen unterschieden, wobei die fünfte Klasse (magenta) äußerst selten vorkam, aber einen sehr hohen Rückstreukoeffizienten hatte.
1 def db_hist(I,b,r):
2 """Input: I ist das Intensitätsbild, b ist die Anzahl der bins, r ist die Limitierung der x-Achse"""
3 A_db=nan_to_num(10*log10(I))
4 h=histogram(A_db,bins=b,range=r,normed=True)
5 return h[0],h[1] # h[0]: y-Achse des Histogramms, h[1]: x-Achse des Histogramms
6
7 # Definition der homogenen Bereiche
8 A_db=10*log10(A[:,:,0])
9 A1=A[295:394,929:997,0]
10 A2=A[198:225,168:197,0]
11 A3=A[863:949,382:464,0]
12 A4=A[846:851,623:629,0]
13 Aice=A[763:771,326:329,0] # vermutlich Eisberge
14
15 b=50
16 r=[-20,5]
17
18 y1,x1=db_hist(A1,b,r)
19 y2,x2=db_hist(A2,b,r)
20 y3,x3=db_hist(A3,b,r)
21 y4,x4=db_hist(A4,b,r)
22 yice,xice=db_hist(Aice,b,r)
23
24 # Histogramm
25 figure(7)
26 plot(x1,y1,'g',x2,y2,'b',x3,y3,'y',x4,y4,'r',xice,yice,'m')
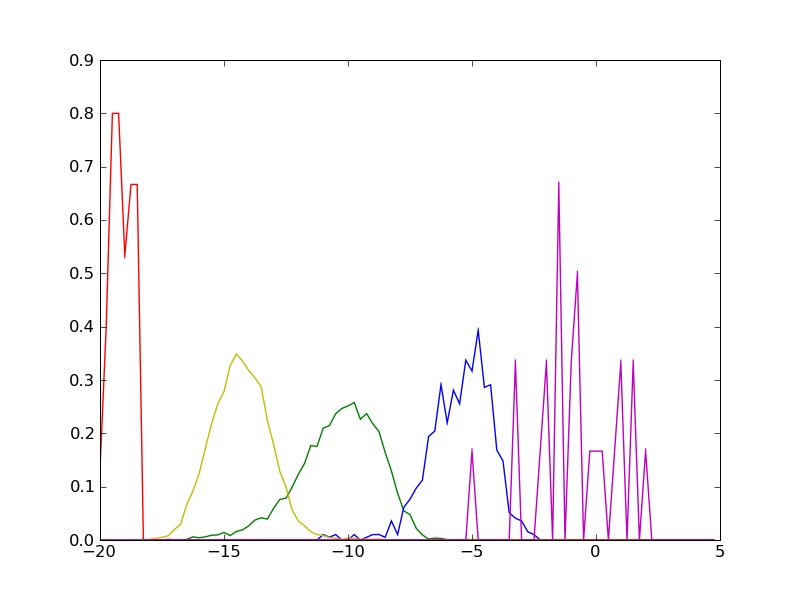
Die Schwellwerte haben wir aus dem Histogramm abgelesen.
1 # Klassifikation
2
3 # Schwellwerte
4 s0=-17.3
5 s1=-12.4
6 s2=-7.88
7 s3=-1.73
8
9 A_class=A_db.copy()
10 A_class[A_db<s0]=0
11 A_class[A_db>s0]=1
12 A_class[A_db>s1]=2
13 A_class[A_db>s2]=3
14 A_class[A_db>s3]=4
15
16 figure(11)
17 imshow(A_class,cm.gist_rainbow,interpolation='nearest',origin='lower')
18 colorbar()
19 show()
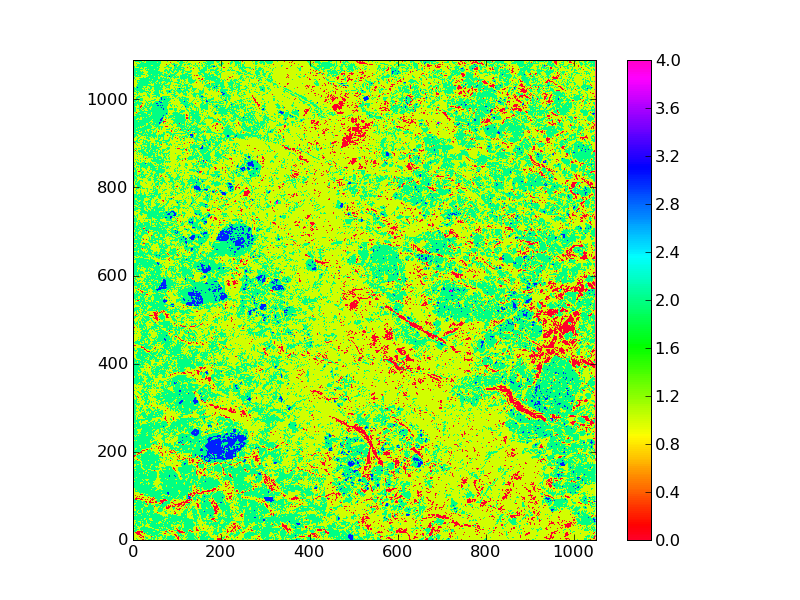
Diese Bilder stimmen noch nicht ganz mit dem überein, was im Programmtext steht. Morgen geht es weiter.
