Besprechung ihrer Aufgaben ergab:
- Einlesen als Teilaufgabe wurde von Lars geloest Gruppe arbeitet direkt mit Bildkoordinaten, welche als Arrayindex dienen
Festlegung der Klasseneinteilung erst bei Bearbeitung der Aufgabe -> Sinnigkeitsentscheid Verwendung von Filtern und Clusterbasierte Merkmalsanalyse Ergebnis: die Zuordnung von Klassen zu Bildkoordinaten (x,y)
Dienstag
Wir haben ein Programm geschrieben, um über ein großes Bild einen Box-Filter laufen zu lassen, der Mittelwert und Standardabweichungen in einem 2-dimensionalen Array zurückgibt.
1 def mean_std_box(I,w):
2 """ Mittelwert über wxw Pixel"""
3 Y,X=I.shape # Einlesen der Bilddimension
4 M=zeros((Y/w,X/w,2),float)
5 a=range(0,Y/w,1)
6 b=range(0,X/w,1)
7 for y in a:
8 for x in b:
9 box=I[y*w:y*w+w-1,x*w:x*w+w-1]
10 M[y,x,0]=mean(box.flatten())
11 M[y,x,1]=std(box.flatten())
12 return M
Weiter haben wir uns damit beschäftigt, das Originalbild mit dem "read_asar_imp" einzulesen und in db-Darstellung auszugeben.
1 # Einlesen des Originalbildes
2 file='/pf/u/u242027/SAR_raw/ASA_IMP_1PNDPA20060617_043346_000000162048_00362_22460_2136.N1'
3 I=read_asar_imp(file)
4
5 # Darstellung des ASAR-Bildes in db (image1.png)
6 w=8 # Größe der "Unterboxen"
7 A=mean_std_box(I,w)
8
9 figure(2)
10 imshow(10*log10(A[:,:,0]),vmin=-20,vmax=0,interpolation='nearest',origin='lower')

Probleme hatten wir bei der Reduzierung des Speckles. Es wurde daher beschlossen diesen Schritt am Mittwoch zunächst in der ganzen Gruppe zu besprechen und dann durchzuführen.
Mittwoch
Wir haben eine Klassifizierung nur über den Mittelwert durchgeführt. Dazu haben wir das Bild in db umgerechnet und uns ein Histogramm erzeugt, bei dem jedoch keine Klassen zu unterscheiden waren. Auch eine Filterung mit dem Lee-Filter und dem Programm smooth hat am Histogramm nicht so viel verändert.
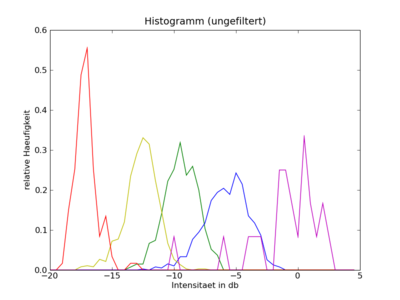
Daher haben wir aus dem gemittelten Bild homogene Bereiche ausgewählt und diese in einem Histogramm geplottet. Insgesamt haben wir fünf Klassen unterschieden, wobei die fünfte Klasse (magenta) äußerst selten vorkam, aber einen sehr hohen Rückstreukoeffizienten hatte.
1 def db_hist(I,b,r):
2 """Input: I ist das Intensitätsbild, b ist die Anzahl der bins, r ist die Limitierung der x-Achse"""
3 A_db=nan_to_num(10*log10(I))
4 h=histogram(A_db,bins=b,range=r,normed=True)
5 return h[0],h[1] # h[0]: y-Achse des Histogramms, h[1]: x-Achse des Histogramms
6
7 # Definition der homogenen Bereiche
8 A_db=10*log10(A[:,:,0])
9 A1=A[295:394,929:997,0]
10 A2=A[198:225,168:197,0]
11 A3=A[863:949,382:464,0]
12 A4=A[846:851,623:629,0]
13 Aice=A[763:771,326:329,0] # vermutlich Eisberge
14
15 b=50
16 r=[-20,5]
17
18 y1,x1=db_hist(A1,b,r)
19 y2,x2=db_hist(A2,b,r)
20 y3,x3=db_hist(A3,b,r)
21 y4,x4=db_hist(A4,b,r)
22 yice,xice=db_hist(Aice,b,r)
23
24 # Histogramm
25 figure(7)
26 plot(x1,y1,'g',x2,y2,'b',x3,y3,'y',x4,y4,'r',xice,yice,'m')
Die Schwellwerte haben wir aus dem Histogramm abgelesen.
1 # Klassifikation
2
3 # Schwellwerte
4 s0=-17.3
5 s1=-12.4
6 s2=-7.88
7 s3=-1.73
8
9 A_class=A_db.copy()
10 A_class[A_db<s0]=0
11 A_class[A_db>s0]=1
12 A_class[A_db>s1]=2
13 A_class[A_db>s2]=3
14 A_class[A_db>s3]=4
15
16 figure(11)
17 imshow(A_class,cm.gist_rainbow,interpolation='nearest',origin='lower')
18 colorbar()
19 show()
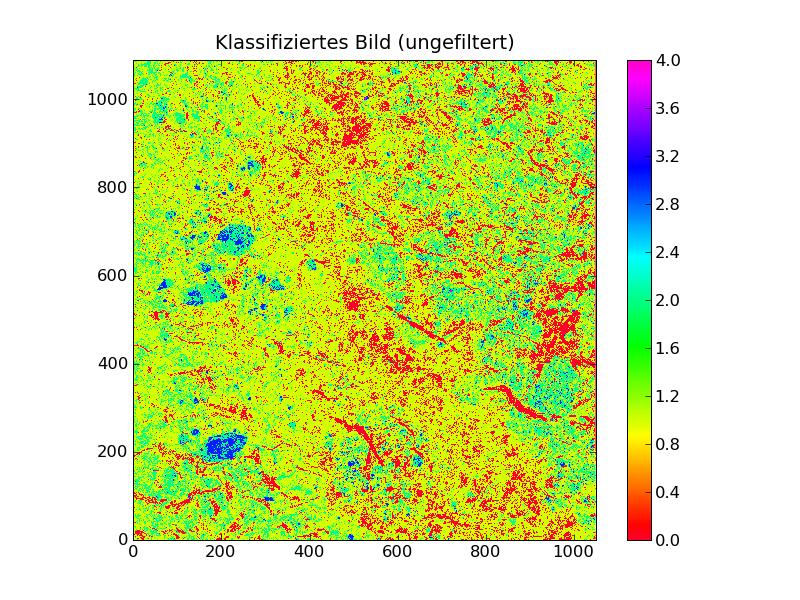
Die Klassifikation ist noch nicht perfekt.
Donnerstag und Freitag
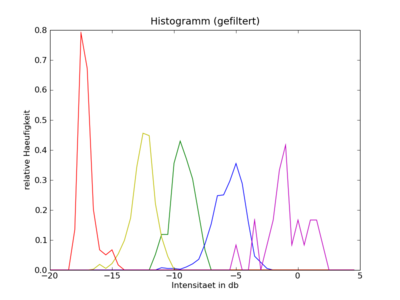
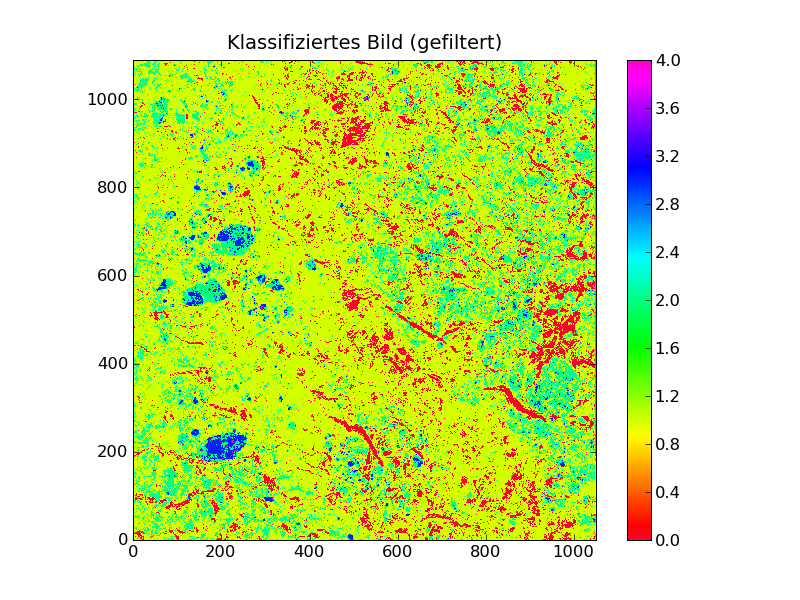
Wir haben die Klassifikation noch etwas optimiert, sodass die wichtigsten Strukturen auf dem klassifizierten Bild erkennbar sind. Wir haben außerdem noch ein Bild erstellt, das zusätzlich einen "smooth"-Filter durchlaufen hat.
Dies ist der Programmtext der endgültigen Version.
1 # Programm zur Klassifikation von Eistypen eines ASAR-Bildes
2
3 from scipy import *
4 from pylab import *
5 from read_asar import *
6 import scipy.stats
7 import os.path
8 import scipy.ndimage as ndi
9
10
11 def mean_std_box(I,w):
12 """ Mittelwert über wxw Pixel"""
13 Y,X=I.shape # Einlesen der Bilddimension
14 M=zeros((Y/w,X/w,2),float)
15 a=range(0,Y/w,1)
16 b=range(0,X/w,1)
17 for y in a:
18 for x in b:
19 box=I[y*w:y*w+w-1,x*w:x*w+w-1]
20 M[y,x,0]=mean(box.flatten())
21 M[y,x,1]=std(box.flatten())
22 return M
23
24 def smooth(I,N):
25 """Box average filter"""
26 kernel=ones((N,N),float32)/(N**2)
27 return ndi.convolve(I, kernel)
28
29 def db_hist(I,b,r):
30 I_db=nan_to_num(10*log10(I))
31 h=histogram(I_db,bins=b,range=r,normed=True)
32 return h[0],h[1]
33
34 def classification(A_db,s):
35 """liefert klassifiziertes Bild aus db-Bild"""
36 # s ist eine Liste mit beliebig vielen Schwellwerten
37 A_cl=A_db.copy()
38 A_cl[A_db<s[0]]=0
39 for i in range(len(s)):
40 A_cl[A_db>s[i]]=i+1
41 return A_cl
42
43 def db_image(A):
44 """rechnet Intensität in dezibel um"""
45 return 10*log10(A)
46
47
48 #*********************************************************************
49 # Einlesen des Originalbildes
50
51 filename='/pf/u/u241110/project/asar_box_1090x1051x2.dat'
52 if not(os.path.exists(filename)):
53 file='/pf/u/u242027/SAR_raw/ASA_IMP_1PNDPA20060617_043346_000000162048_00362_22460_2136.N1'
54 I=read_asar_imp(file)
55 w=8 # Größe der "Unterboxen"
56 A=mean_std_box(I,w)
57 A.tofile(filename)
58 else:
59 A=reshape(fromfile(filename),(1090,1051,2))
60
61 #*********************************************************************
62 # Darstellung des ASAR-Bildes in db (image1.png)
63
64 #figure(1)
65 imshow(db_image(A[:,:,0]),vmin=-20,vmax=0,interpolation='nearest',origin='lower')
66 gray()
67 colorbar()
68 title('ASAR-Bild in db')
69
70 #*********************************************************************
71 # Streudiagramm
72
73 figure(8)
74 plot(A[:,:,0].flatten(),A[:,:,1].flatten(),'.')
75 xlabel('Mittelwert')
76 ylabel('std')
77
78 #*********************************************************************
79 # Histogramme
80
81 # Festlegung der homogenen Flächen
82 A_db=db_image(A[:,:,0])
83 A_homogen=(A[365:380,976:994,0],A[198:225,168:197,0],A[830:860,55:80,0],A[291:298,877:894,0],A[763:771,326:329,0])
84
85 # gefiltertes Bild
86 A_sm=smooth(A[:,:,0],2)
87 A_db_sm=db_image(A_sm)
88 A_homogen_sm=(A_sm[365:380,976:994],A_sm[198:225,168:197],A_sm[830:860,55:80],A_sm[291:298,877:894],A_sm[763:771,326:329])
89
90 #b=bins, r=Länge der X-Achse im Histogramm
91 b=50
92 r=[-20,5]
93
94 # Erzeugung des Histogramms
95 y=zeros((b,len(A_homogen)),float)
96 x=zeros((b,len(A_homogen)),float)
97 for i in range(len(A_homogen)):
98 y[:,i],x[:,i]=db_hist(A_homogen[i],b,r)
99
100 y_sm=zeros((b,len(A_homogen)),float)
101 x_sm=zeros((b,len(A_homogen)),float)
102 for i in range(len(A_homogen_sm)):
103 y_sm[:,i],x_sm[:,i]=db_hist(A_homogen_sm[i],b,r)
104
105 # graphische Darstellung
106 cl=['g','b','y','r','m']
107
108 figure(2)
109 for i in range(len(A_homogen)):
110 plot(x[:,i],y[:,i],cl[i])
111 hold(True)
112
113 title('Histogramm')
114 xlabel('Intensitaet in db')
115 ylabel('relative Haeufigkeit')
116
117 figure(3)
118 for i in range(len(A_homogen_sm)):
119 plot(x_sm[:,i],y_sm[:,i],cl[i])
120 hold(True)
121
122 title('Histogramm (gefiltert)')
123 xlabel('Intensitaet in db')
124 ylabel('relative Haeufigkeit')
125
126 #*************************************************************************
127 # Klassifikation
128
129 # Schwellwerte
130 s=[-15.1,-11,-7.56,-1.98]
131 A_class=classification(A_db,s)
132
133 # gefiltertes Bild
134 s=[-15.2,-11,-7.83,-3.09]
135 A_class_sm=classification(A_db_sm,s)
136
137 # graphische Darstellung
138 figure(4)
139 imshow(A_class,cm.gist_rainbow,interpolation='nearest',origin='lower')
140 colorbar()
141 title('Klassifiziertes Bild (ungefiltert)')
142 #A_class.tofile('asar_class_1090x1051.dat')
143
144 figure(5)
145 imshow(A_class_sm,cm.gist_rainbow,interpolation='nearest',origin='lower')
146 colorbar()
147 title('Klassifiziertes Bild (gefiltert)')
148 #A_class_sm.tofile('asar_class_filtered_1090x1051.dat')
149
150 #**********************************************************************
151 show()
Die klassifizierten Arrays sind unter /pf/u/u241110/project/asar_class_1090x1051.dat bzw. .../asar_class_filtered_1090x1051.dat